Tendo recentemente construído muitas árvores filogenéticas com o módulo TreeConstruction do pacote Phylo da Biopython, fui solicitado a substituir os rótulos das pontas dos ramos pelos logotipos de sequência correspondentes (que tenho na mesma pasta). Achei que seria mais eficiente fazer um código para gerar as árvores de logotipo automaticamente, já que teria que fazer muitos deles.
A primeira ideia que tive foi ver se as funções usadas para construir a árvore tinham um argumento para substituir os rótulos das pontas do galho ou removê-los, que não consegui encontrar. Portanto, removi os rótulos de ponta de ramificação, definindo seu tamanho de fonte para 0: (a seguir está o código para construir a árvore)
# Módulos para construir a árvore de Bio.Phylo.TreeConstruction import DistanceCalculator, DistanceTreeConstructorfrom Bio.Phylo import drawfrom Bio import Phylo, AlignIOimport subprocessimport matplotlibimport matplotlib.pyplot as pltalignment = AlignIO.read ('MotifSeqAligned.fasta', 'fasta') # lendo o alinhamento filecalculator = calculadora_docalador = DistanceCalculator ') (alinhamento) # distance matrixconstructor = DistanceTreeConstructor () tree = constructor.nj (dm) # construir com o algoritmo de junção de vizinho uma árvore de dmPhylo.write (tree, 'TreeToCutOff.nwk', 'newick') plt.rc ('font' , tamanho = 0) # controla os tamanhos de texto padrão #AQUI ESTÁ A CONFIGURAÇÃO PARA QUE ME PERMITE OCULTAR A DICA DE RAMO LABELSplt.rc ('eixos', tamanho da fonte = 14) # tamanho da fonte dos eixos titleplt.rc ('xtick', tamanho da etiqueta = 10) # tamanho da fonte do tick labelsplt.rc ('ytick', labelsize = 10) # fontsize do tick labelsplt.rc ('figure', title = 18) # fontsize da figura entituladaraw (tree, do_show = False) plt.savefig ("TreeToCutOff.svg", format = 'svg', dpi = 1200) Deste código eu poderia obter a árvore:
Como não sei como obter as coordenadas y dos ramos para adicionar os logotipos um por um, construí uma coluna de logotipos com matplotlib, que pretendia colar na árvore em python. O código para construir a coluna de logotipos é o seguinte:
#Extract filename from newicknewickFile = open ("TreeToCutOff.nwk", 'r'). Read () OrderLogos = ["{} .eps ".format (i) for i in re.split ('(\ W)', newickFile) if" Profile "in i] #Inicialize a figurafig = plt.figure () # Adicione cada imagem uma após a outra em a ordem certa para i, arquivos em enumerar (OrderLogos): img1 = mpimg.imread (arquivos) ax1 = fig.add_subplot (len (OrderLogos), 1, 1 + i) ax1.imshow (img1) ax1.set_xticks ([]) ax1.set_yticks ([]) # plt.show () plt.savefig ("RowsOfLogos.svg", format = 'svg', dpi = 1200) plt.clf () plt.cla () Tendo minha árvore e a coluna de logotipos em .svg ou .png, não consegui encontrar nenhuma maneira de empilhá-los corretamente. Minha primeira ideia era usar a biblioteca svgutils que parecia ser fácil de manusear, com o seguinte código: (retirado dos tutoriais de svgutils)
import svgutils.transform asg # Assemble # create new SVG figurefig = sg.SVGFigure ("14cm", "14cm") # load Figuresfig1 = sg.fromfile ('TreeToCutOff.svg') fig2 = sg.fromfile ('RowsOfLogos.svg') # get the plot objectsplot1 = fig1.getroot ( ) plot2 = fig2.getroot () plot2.moveto (280, 100, escala = 0,05) # anexar gráficos e rótulos a figurefig.append ([plot1, plot2]) Mas o problema com a saída foi que o fundo da coluna de logotipos era branco e, portanto, eu estava colando uma enorme imagem branca com uma fina coluna de logotipos na árvore. E não consegui encontrar uma maneira de cortar a coluna de logotipos com svgutils. Tentei o módulo Imagem do pacote PIL para construir uma árvore de logotipos a partir de arquivos .png, mas não consegui ver a árvore usada como plano de fundo após colar a coluna de logotipos.
Pode haver uma maneira de fazer o que pretendo com matplotlib (que seria empilhar 2 arquivos .png e colocar os logotipos o tempo todo na mesma distância), mas não consegui resolver .
Alguém sabe qual a melhor solução é fazer uma árvore de logotipos (como na imagem a seguir que eu só poderia construir manualmente com o inkscape) com bibliotecas python, permitindo automatizar o processo sem ter que se adaptar o código depende do número de ramos?
A seguir está um subconjunto de "MotifSeqAligned.fasta" contendo as sequências alinhadas usadas para construir as árvores:
>ProfileCluster0.meme --- SSNDTTTCCAGGAAD->ProfileCluster1.memeYBNRD --- TTCYYGGAAT->ProfileCluster10.meme- VDKDWT com 3 logotipos correspondentes: 
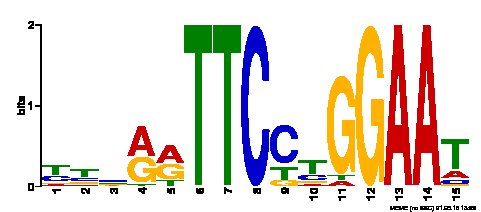
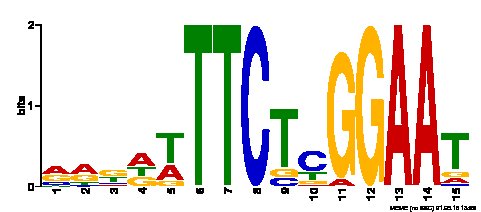
O "MotifSeqAligned.fasta" completo e todos os logos.eps (como eu os usei em vez de .png que é o formato solicitado pelo fórum) podem ser encontrados aqui.